NEJM | 陆林宇团队揭示无精症患者中MOV10L1基因变异与piRNA加工缺陷
全球约有15%的夫妇不育,其中男性因素约占一半。无精症是导致男性不育的重要原因,其中非梗阻性无精症(nonobstructive azoospermia)表现为睾丸中精子发生障碍,治疗手段非常有限。部分非梗阻性无精症由遗传因素造成,包括染色体数目或结构异常与基因变异等。piRNA(Piwi-interacting RNA)是一类长度约为24-32 nt的小RNA,在生殖细胞中特异表达,对转座子的调控至关重要。piRNA的成熟需要对其前体的5’及3’端进行加工。在各类模式动物中,piRNA加工缺陷通常导致生殖细胞发育障碍。PNLDC1是一个参与piRNA 3’端加工的核酸外切酶。2021年8月发表在New England Journal of Medicine的一篇文章报道了非梗阻性无精症患者中的PNLDC1基因变异与piRNA加工缺陷[1]。

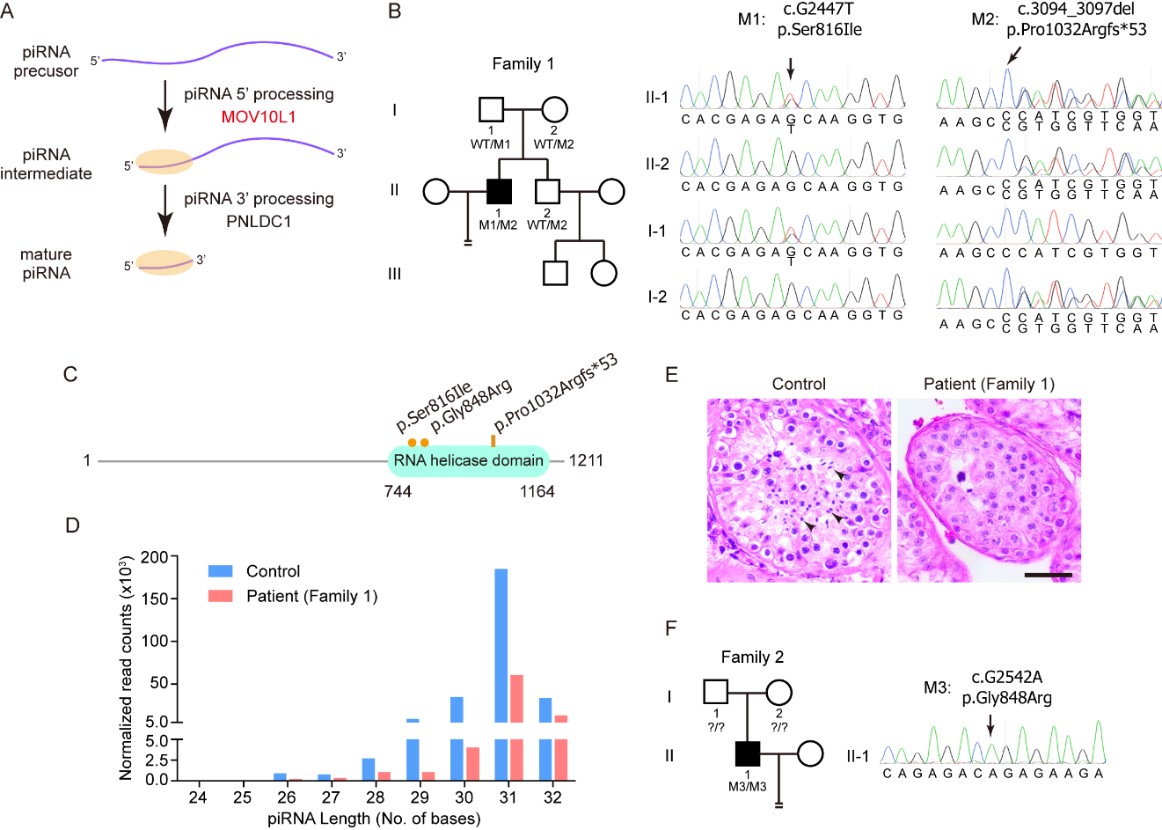
MOV10L1是一个参与piRNA 5’端加工的RNA解旋酶[2, 3],位于PNLDC1的上游。2022年4月28日,浙江大学转化医学研究院/医学院附属妇产科医院陆林宇课题组在New England Journal of Medicine发表题为Defective piRNA Processing and Azoospermia的通讯,报道了非梗阻性无精症患者中的MOV10L1基因变异与piRNA加工缺陷。
该研究招募了一名在浙江大学医学院附属妇产科医院就诊的非梗阻性无精症患者,其睾丸穿刺样品的病理显示生精阻滞,减数分裂不能完成。研究进一步提取了患者、其可育兄弟、其父母四人的基因组DNA并通过全外显子组测序分析发现,患者携带分别来自父母的两个MOV10L1基因变异:p.Ser816Ile和p.Pro1032Argfs*53。研究通过小RNA测序发现,患者睾丸穿刺样品中的piRNA数量显著降低。由于这两个基因变异均位于MOV10L1的RNA解旋酶结构域,两者可能均破坏了MOV10L1的RNA解旋酶活性。这一发现与小鼠中MOV10L1酶活突变导致piRNA加工缺陷及减数分裂阻滞的现象高度吻合[4, 5]。根据ACMG遗传变异分类标准与指南[6],这两个基因变异分别为“可能致病”及“致病”。研究进一步利用中南大学谭跃球团队的414名非梗阻性无精症或严重少精症患者样品进行筛查,发现其中一名患者携带一个纯合的MOV10L1基因变异:p.Gly848Arg。这个基因变异也位于RNA解旋酶结构域,可能也破坏了MOV10L1的RNA解旋酶活性。根据ACMG遗传变异分类标准与指南,这个基因变异为“意义不明确”,其致病性值得后续研究。该研究揭示了非梗阻性无精症患者中的MOV10L1基因变异,并与前期研究呼应,共同提示piRNA加工缺陷可能是导致男性非梗阻性无精症的重要原因。
浙江大学转化医学研究院/医学院附属妇产科医院陆林宇为通讯作者,浙江大学医学院附属妇产科医院李乐军为第一作者,中南大学谭跃球为重要的合作者。浙江大学医学院附属妇产科医院刘一丹全面负责了患者家系的测序及遗传分析。本研究还得到了浙江大学医学院附属妇产科医院李鹏,中南大学胡童谣、何文斌的帮助。同时感谢各位匿名的患者及其家庭对本研究的支持。
陆林宇为浙江大学转化医学研究院全职PI,同时双聘于浙江大学医学院附属妇产科医院。课题组主要研究方向为DNA损伤修复与生殖细胞发育,同时也研究DNA损伤修复维持基因组稳定性的分子机制。课题组主要利用细胞与小鼠模型进行研究,并与临床紧密合作,详情请见课题组主页:https://person.zju.edu.cn/lulinyu。陆林宇课题组长期招收博士后与科研助理,欢迎申请。有意者可邮件联系:lulinyu@zju.edu.cn。
原文链接:
https://www.nejm.org/doi/full/10.1056/NEJMc2116008
参考文献:
1.Nagirnaja, L., et al., Variant PNLDC1, Defective piRNA Processing, and Azoospermia. N Engl J Med, 2021. 385(8): p. 707-719.
2.Zheng, K., et al., Mouse MOV10L1 associates with Piwi proteins and is an essential component of the Piwi-interacting RNA (piRNA) pathway. Proc Natl Acad Sci U S A, 2010. 107(26): p. 11841-6.
3.Frost, R.J., et al., MOV10L1 is necessary for protection of spermatocytes against retrotransposons by Piwi-interacting RNAs. Proc Natl Acad Sci U S A, 2010. 107(26): p. 11847-52.
4.Vourekas, A., et al., The RNA helicase MOV10L1 binds piRNA precursors to initiate piRNA processing. Genes Dev, 2015. 29(6): p. 617-29.
5.Fu, Q., et al., Mutations in the MOV10L1 ATP Hydrolysis Motif Cause piRNA Biogenesis Failure and Male Sterility in Mice. Biol Reprod, 2016. 95(5): p. 103.
6.Richards, S., et al., Standards and guidelines for the interpretation of sequence variants: a joint consensus recommendation of the American College of Medical Genetics and Genomics and the Association for Molecular Pathology. Genet Med, 2015. 17(5): p. 405-24.


