Genome Biology丨谢安勇教授团队发现CRISPR靶点滞留影响靶点DNA修复途径选择
CRISPR/Cas9基因编辑依赖于细胞内源DNA双链断裂(double strand break, DSB)修复机器实现,因此也受同样的DSB修复途径选择调控机制控制。然而,Cas9诱导产生DSB的过程及产生的DSB构象非常特别,其修复调控机制也独具特色,特别是DSB产生后CRISPR/Cas9仍能在靶点滞留相当长的一段时间,阻碍DSB末端的暴露以及随后的DNA损伤应答与修复1,2。我们于是推测,CRISPR/Cas9与断裂靶点的滞留会影响DSB修复途径选择,增添了一层DSB修复途径选择调节3。2022年8月1日,浙江大学转化医学研究院/浙江大学医学院附属邵逸夫医院谢安勇教授团队在Genome Biology杂志上发表了题为“Target residence of Cas9-sgRNA influences DNA double strand break repair pathway choices in CRISPR/Cas9 genome editing”的研究论文,揭示了CRISPR/Cas9末端滞留对靶点DSB修复途径选择的影响与机制。

在哺乳动物细胞中,DSB主要由同源重组(homology-directed repair,HDR)和非同源末端连接(non-homologous end joining,NHEJ)两条途径修复。NHEJ根据参与元件的不同又可以分为经典型NHEJ(c-NHEJ)和替代型NHEJ(a-EJ)。其中c-NHEJ负责完成大部分的NHEJ修复,需要DNA-PKcs、Ku70/Ku80和XRCC4/DNA ligase 4等NHEJ核心因子的共同参与,可以高效、精准地修复Cas9诱导的平末端DSB4,5。而a-EJ则是在缺乏其中任一个核心NHEJ因子参与的情况下进行DSB修复,精准度差。作者通过研究同一基因片段附近不同Cas9编辑位点的DSB修复行为发现,不同靶点c-NHEJ修复途径的依赖性存在显著差异,甚至有的位点完全不涉及c-NHEJ。进一步研究发现,c-NHEJ途径的依赖性与Cas9-sgRNA复合物的滞留能力密切相关。体外实验显示,改变sgRNA序列使其与靶DNA产生碱基错配可以加速Cas9-sgRNA复合物从靶DNA上解离,缩短靶点滞留时间。在小鼠胚胎干细胞内,当有的靶点不依赖于c-NHEJ进行DSB修复时,如果使用sgRNA-DNA碱基错配、sgRNA截短和Cas9变体降低Cas9-sgRNA复合物的靶点结合能力,同一靶点的CRISPR/Cas9基因编辑将更偏向于利用c-NHEJ进行DSB修复,而抑制c-NHEJ将更显著地提高HDR和a-EJ效率。同时也解释了为什么在有的靶点(即c-NHEJ没有参与的靶点),利用NHEJ抑制剂并不能提高HDR的效率。
相似地,由于碱基错配,脱靶位点CRISPR/Cas9滞留能力较低,脱靶位点的DSB修复会有更多的c-NHEJ参与。同时因为脱靶位点CRISPR/Cas9的识别和结合弱,被再切的概率低,因此脱靶位点的精准修复效率高而突变通常较少。然而,抑制c-NHEJ将提高脱靶位点的a-EJ的利用率,增加突变。事实上,该研究发现,在利用抑制c-NHEJ的方式提升HDR介导的基因敲进时,不可避免地加重了脱靶效应。
接下来,作者分析靶点滞留与基因转录和DNA复制的相互作用如何影响DSB修复途径选择。DNA切割后滞留的Cas9-sgRNA可能遭遇DNA转录与复制,并从靶点解离,从而暴露出DNA断裂末端。然而,Cas9-sgRNA复合物诱导DSB时,c-NHEJ的参与修复的程度与催化活性失活的dCas9-sgRNA在同一位点的转录阻断能力并没有相关性。Cas9-sgRNA复合物在靶点的转录解离也不影响该靶点的DSB修复的c-NHEJ参与度。这说明,转录解离Cas9-sgRNA后并不影响CRISPR靶点的修复途径选择。相反,与DNA复制偶联的Cas9-sgRNA靶点解离减少了c-NHEJ的参与度而提高了HDR。于是,降低Cas9-sgRNA靶点滞留可以降低与DNA复制叉碰撞,使得Cas9-sgRNA更偏向于自然解离,提高了c-NHEJ修复途径的选择,降低了HDR。此外,DNA切割后,与DNA复制叉碰撞将解离Cas9-sgRNA,产生三末端DSB,其中两条新合成的姐妹染色单体应该有一定几率进行连接,形成巨型回文染色体(Palindromic chromosome)6,7。利用PCR和测序,作者在编辑产物中检测到这种回文染色体产物,并证实这种产物的形成与DNA复制偶联。
该研究揭示了Cas9-sgRNA末端滞留影响靶点DSB修复途径选择(图1),也提出了下一个问题:在应用CRISPR/Cas9基因编辑时,我们是否能够更有效地操纵和利用CRISPR/Cas9的靶点滞留?事实上,利用Cas9单链缺刻酶靶点滞留的特点,联合DNA复制,我们模拟细胞内DNA单链断裂的产生及复制偶联单末端DSB的转换,发现DNA单链断裂及其修复异常是BRCA1缺陷肿瘤特征性突变模式产生的一个主要来源与机制8。
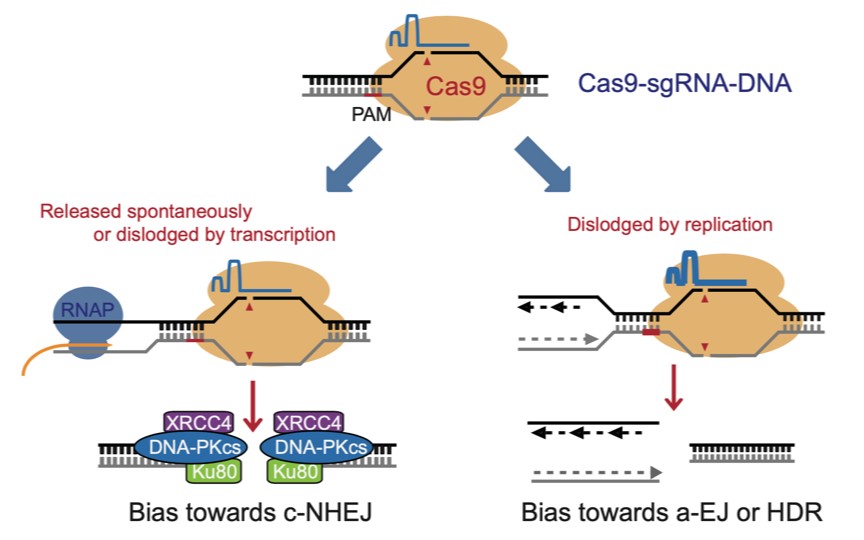
图1. CRISPR/Cas9靶点滞留对靶点DSB修复途径选择的影响
该论文通讯作者是浙江大学医学院附属邵逸夫医院和浙江大学医学院转化医学研究院谢安勇教授。刘嗣诚和冯依力博士是本文第一作者,硕士研究生孙秀娜也做了大量的工作。本课题得到了国家自然科学基金面上项目和浙江省自然科学基金项目的支持。谢安勇教授团队致力于肿瘤基因组不稳定性和CRISPR基因编辑技术机制与改良的研究,已先后在Mol Cell,Nat Struct Mol Biol,Nat Commun,Genome Biol,Nucleic Acids Res等一系列期刊发表学术论文。
参考文献
1. Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C. & Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature 507, 62–67 (2014).
2. Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L. & Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nat. Biotechnol. 34, 339–344 (2016).
3. Feng, Y., Liu, S., Chen, R. & Xie, A. Target binding and residence: a new determinant of DNA double-strand break repair pathway choice in CRISPR/Cas9 genome editing. J Zhejiang Univ Sci B 22, 73–86 (2021).
4. Guo, T. et al. Harnessing accurate non-homologous end joining for efficient precise deletion in CRISPR/Cas9-mediated genome editing. Genome Biol. 19, 170 (2018).
5. Yin, J. et al. Cas9 exo-endonuclease eliminates chromosomal translocations during genome editing. Nat Commun 13, 1204 (2022).
6. Umbreit, N. T. et al. Mechanisms generating cancer genome complexity from a single cell division error. Science 368, eaba0712 (2020).
7. Tanaka, H. & Watanabe, T. Mechanisms Underlying Recurrent Genomic Amplification in Human Cancers. Trends Cancer 6, 462–477 (2020).
8. Feng, Y.-L. et al. DNA nicks induce mutational signatures associated with BRCA1 deficiency. Nat Commun 13, 4285 (2022).


